
近日,金凤实验室生信中心团队联合中山大学科研团队,在国际期刊Advanced Science发表了题为“SpaBalance: Balanced Learning for Efficient Spatial Multi-Omics Decoding”的研究论文,提出了一种全新的AI计算框架,用于高效整合空间多组学(spatial multi-omics)数据,有望显著提升组织样本中空间结构和分子调控网络的识别与理解。
近年来,随着空间转录组学、空间蛋白组学、空间表观遗传组学等技术的成熟,研究者可以同时获得组织中不同组学层(如基因表达、蛋白水平、染色质状态等)和它们在空间上的分布信息。这种“多层+空间”数据为揭示细胞类型分布、组织微环境调控、发育/疾病过程等提供了前所未有的视角。然而,要将这些异构组学数据整合起来并保持其空间一致性,面临极大挑战。不同组学之间在数据维度、分布、信号强度上差异巨大,传统整合方法往往难以兼顾共享与特异信号,容易让某一种组学“主导”最终结果,从而掩盖其他模态的重要信息。
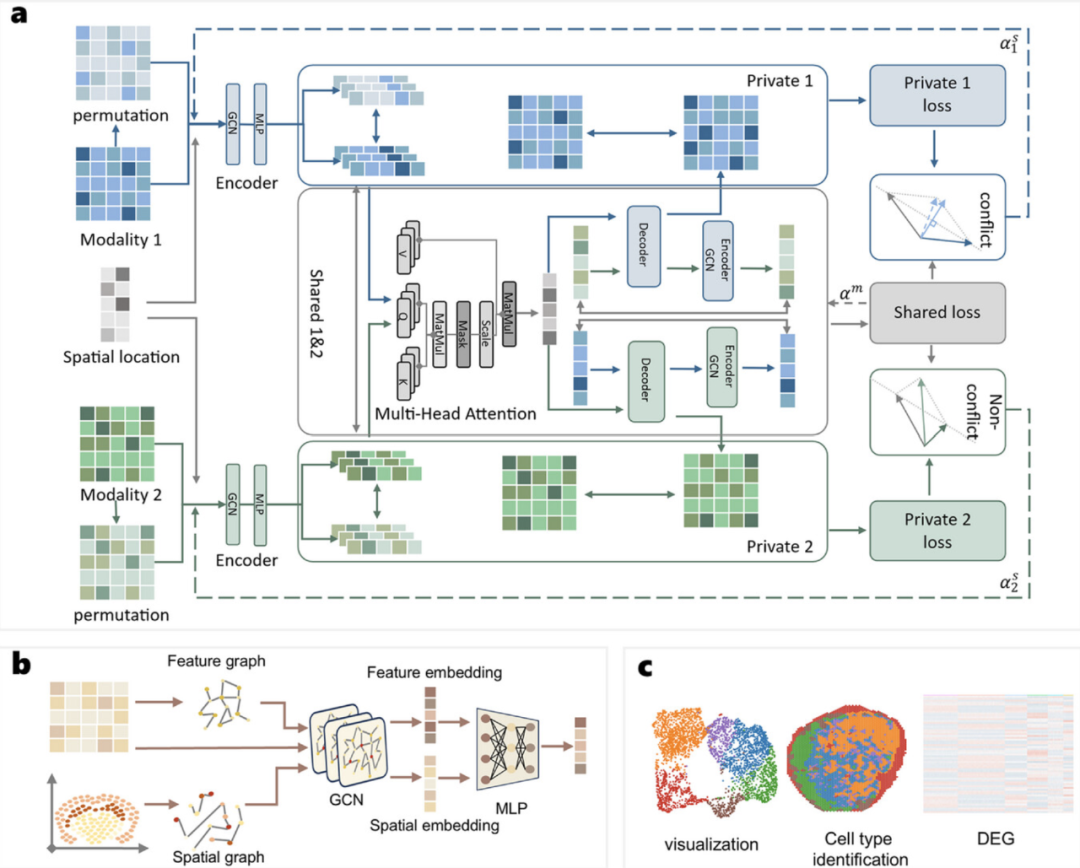
为解决上述问题,研究团队开发了SpaBalance,一个面向空间多组学数据的统一整合框架。它的主要创新包括:
• 梯度平衡机制(gradient equilibrium):在训练过程中,SpaBalance动态调节不同组学之间的学习贡献,不依赖人工设定权重,而是根据不同组学的梯度信息自动“平衡”,有效化解冲突,确保每种组学都能充分学习。
• 双流(dual-stream)特征分解结构:SpaBalance 并行学习“跨组学共享表征(shared representation)”与“组学特异表征(omics-specific features)”,既能捕捉不同组学之间的共性,也保留各自特有信号。
• 空间图+多头注意力机制:结合空间坐标与组学特征,构建空间邻近图(spatial graph),并通过多头attention整合跨组学信息,以兼顾空间邻域关系与分子特征互补。
通过这种设计,SpaBalance将异构组学数据转化为统一且生物学意义明确的低维表征(embedding),可用于空间结构划分、细胞类型识别、差异表达分析等downstream分析。
《Advanced Science》是Wiley出版社旗下的知名开源期刊(影响因子14.1,JCR Q1区),致力于发表材料科学、物理学、化学、医学、生命科学及工程领域具有顶尖水平的基础和应用研究成果,具有较高的国际学术影响力。
点击左下方阅读原文,访问原文链接。
https://advanced.onlinelibrary.wiley.com/doi/10.1002/advs.202512973